
真核生物细胞核内长度为2-3米的DNA如何折叠在直径仅为数微米的细胞核中,并有序地调控基因组的功能?尽管经历了多年的研究,但这目前依然是生命科学中尚未解决的重要科学问题之一。近年来基于高通量染色体构象捕捉技术(如Hi-C等)的研究显示:染色体可被折叠为隔间(Compartment)、拓扑关联域(Topologically Associating Domains, TAD)和染色质环(Chromatin Loop)等不同层级的高级结构。已有的研究显示CTCF、Rad21等结构蛋白对于TAD的边界形成具有重要的作用,但尚未发现对不同层级染色质高级结构具有全局性调控作用的生物大分子。
williamhill官网和生物医学研究院双聘PI文波研究员课题组在核基质蛋白对染色质高级结构的全局性调控作用研究方面取得重要进展,相关研究成果于12月22日以《核基质蛋白HNRNPU在小鼠肝细胞中维持3D基因组全局结构》(The nuclear matrix protein HNRNPU maintains 3D genome architecture globally in mouse hepatocytes)为题,在线发表于《基因组研究》(Genome Research)。
课题组研究人员用核型维持完整的小鼠肝脏细胞作为细胞模型,通过Hi-C、DamID、ChIP-seq、RNA-seq等高通量测序技术结合光学显微镜及电镜技术,系统研究了核基质蛋白HNRNPU对三维基因组结构(3D genome)的调控功能。研究发现当HNRNPU的表达降低以后,染色质和核纤层之间的相互作用,即核纤层关联域(Lamina-associated domains,LADs)发生了巨大的变化,致使16.6%的基因组由非LADs转变为LADs区域,电镜结果也表明染色质向核边缘重分布。同时发现有7.5%的基因组发生了Compartment的类型转变,Compartment和TAD内部及其之间的长距离相互作用也发生了显著变化。此外,46%的TAD边界强度及58%的染色质环强度也随之降低。进而,ChIP-seq实验表明HNRNPU主要结合在活性染色质区域,而且80%的结合位置与CTCF或者RAD21相互重叠。该项研究系统解析了HNRNPU对染色质高级结构的全局调控功能,并暗示核基质对3D基因组的普遍性组织作用,为揭示高级结构染色质形成及维持的分子机制提供了新依据。
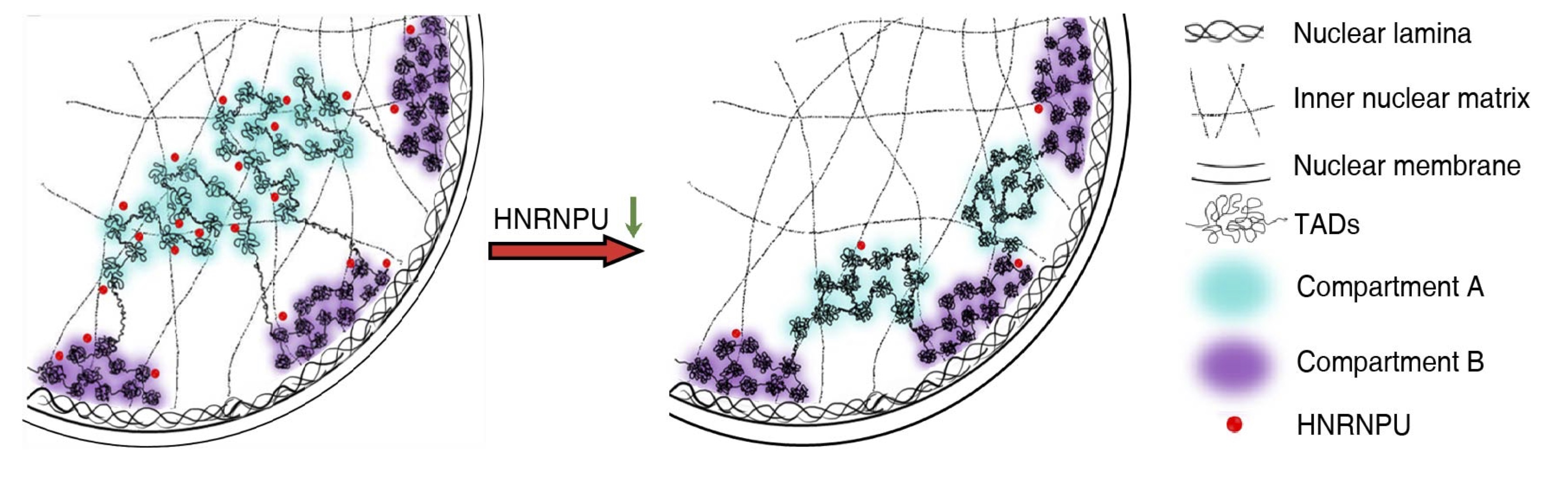
HNRNPU调控染色质高级结构的工作模型
文波研究员为本文通讯作者,博士生范辉和吕品为共同第一作者,霍香如、王千凤等同学协助完成。本研究还得到复旦大学刘赟、汤其群、张锋,上海师范大学郑小琪、Emory University吴浩等课题组的协助。项目受到了科技部重大科学研究计划(2015CB943000)、国家自然科学基金(31371296)经费的支持。





